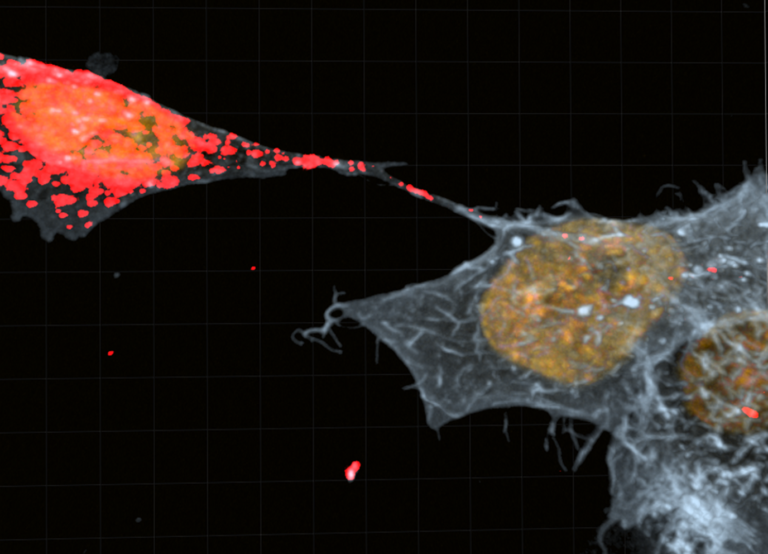
Des chercheurs du Laboratoire IBISC (Informatique, bioinformatique et systèmes complexes, Université d’Evry Paris-Saclay) ont mis au point GraphGONet, un algorithme d’intelligence artificielle qui fournit un diagnostic médical accompagné de l’explication biologique.
L’IA et les cliniciens parlent aujourd’hui la même langue. Grâce à un modèle d’apprentissage profond (Deep learning), trois chercheurs du Laboratoire Ibisc ont conçu GraphGONet, un réseau de neurones virtuels qui associe prédiction et explication. Victoria Bourgeais, Farida Zehraoui et Blaise Hanczar ont publié le modèle dans la revue Bioinformatics du 10 mars 2022.
Les connaissances biologiques des médecins ont été intégrées dans ce réseau de neurones pour que le modèle soit par essence interprétable, en se basant sur la base de connaissances « Gene Ontology », qui uniformise les connaissances biologiques actuellement disponibles sur les gènes et leurs fonctions biologiques. Chaque élément (fonction, mécanisme…), appelé « terme GO », est décrit dans un vocabulaire et des règles standardisées, et codifié. Au moment de livrer sa prédiction, GraphGONet explique donc sur quelles fonctions biologiques il s’appuie.
Jusqu’ici, la plupart des algorithmes utilisés en IA ne livraient pas leurs “recettes” aux équipes médicales. Sans comprendre pourquoi la machine leur livrait tel ou tel diagnostic, une part de doutes subsistait. Avec GraphGONet, chercheurs et cliniciens ont l’assurance que les prédictions reposent bien sur des caractéristiques médicales fiables.
“Les données dont la communauté scientifique dispose aujourd’hui sur les gènes sont très compliquées. Le recours à l’IA est indispensable, en utilisant des méthodes de deep learning. Notre réseau de neurones fait des prédictions capitales sur le patient à partir de l’activité de ses gènes, mais il est essentiel que les cliniciens comprennent comment fonctionne le modèle sinon ils ne l’utiliseront pas”, Blaise Hanczar, co-responsable de l’axe IA du laboratoire IBISC.
En ingérant les données d’expression génique des patients, GraphGONet permet non seulement de déterminer le type de cancer mais aussi d’établir un premier pronostic et de prédire la réponse du patient à tel ou tel traitement. Un pas supplémentaire vers la médecine personnalisée. Un an de recherche a été nécessaire à la conception de ce programme, aujourd’hui libre et disponible sur GitLab.
A terme, les chercheurs espèrent intégrer d’autres sources de connaissances biologiques, telles que les voies métaboliques.